GSL Biotech SnapGene是一款专业的分子生物学软件,这款软件提供了设计引物,酶切位点,氨基酸翻译,密码子优化,序列本地或者网上blast比对等功能,还可以搜索和自定义feature和导入之前的所有引物,功能十分强大!
功能特色:
In-Fusion克隆:一种多功能,多功能的方式,可创建无边界基因连接。 SnapGene是第一个模拟这种方法的软件。只需选择您想要混合的DNA片段,程序就会设计它。
GibsonAssembly:许多研究人员正在将Gibson装配转换为质粒而不使用限制酶。 DNA片段通过PCR连接以干扰。
PCR和诱变:引物设计后,它们可用于常规PCR克隆,共PCR扩增或诱变。得到的DNA序列文件可立即用于进一步操作。
自动文档:自动记录模拟项目中的步骤。每次编辑或模拟序列时,此方法都会自动记录在图形历史记录中。模拟DNA的结构后,您可以使用历史作为实验方案。
琼脂糖凝胶电泳:使用先进的算法创建逼真的琼脂糖模拟。有限部分以三种模拟凝胶格式,数字列表和序列图显示。您可以使用此模拟凝胶来规划诊断约束或将图像与预测模式进行比较。
使用步骤:
首先介绍Snapgene的主要界面:

最下方的视图栏:
(点击下方的图标按钮可以进行相互切换)
最上方的菜单栏:
对应的功能如下:
Map:显示图谱
Sequence:显示序列信息
File:打开、建立、保存相关文件等
Edit:编辑功能,包括复制选中一些序列等
View:切换几个视图等
Enzymes:显示、选择酶切位点等
Feature:显示、增加(命名)一些片段等
Primers:添加引物等Action:插入融合一些片段
Tool:模拟电泳、序列比对、查看DNA分子量、查询简并密码子、氨基酸等
然后我们介绍这个软件的四个视图:
视图一:Map
直接打开任一质粒图谱(以pUC57为例),点击Map按钮,得到如下界面:

 :显示酶切位点。点击该按钮,得到如下界面。
:显示酶切位点。点击该按钮,得到如下界面。

 :显示质粒图谱的开放阅读框及转录方向。点击该按钮,得到如下界面:
:显示质粒图谱的开放阅读框及转录方向。点击该按钮,得到如下界面:

 :显示ORF的片段大小、GC%值等一些信息。点击该按钮,得到如下界面:
:显示ORF的片段大小、GC%值等一些信息。点击该按钮,得到如下界面:

视图二:Sequence
点击Sequence按钮,得到如下界面:

 :显示编码的氨基酸序列。点击该按钮可以切换氨基酸的全称和缩写。
:显示编码的氨基酸序列。点击该按钮可以切换氨基酸的全称和缩写。
 :点击该按钮,选择All6 frames,可以得到所以所有序列编码氨基酸的情况。
:点击该按钮,选择All6 frames,可以得到所以所有序列编码氨基酸的情况。
视图三:Enzymes
点击Enzymes按钮,得到如下界面,可以看到不同酶切位点在序列中位置:

视图四:Features
点击Features按钮,得到如下界面,显示一些常用元件(含元件的位置、大小、功能等信息):

这次我们所讲的这些功能,主要是在最上方的菜单栏进行操作完成:
一、批量添加不同的酶切位点
1.点击菜单栏Enzymes→ChooseEnzymes,可以有选择的显示不同的酶切位点。
2.点击RemoveAll,清空右侧的内切酶,在左侧框内选择需要显示的内切酶进行添加。

二、对片段进行命名
1.在sequence视图,选中需要命名的序列,点击菜单栏Feature→Add Feature,弹出以下窗口:

Feature:给该片段命名。
Type:选择该片段的类型,点击右侧箭头选择不同的阅读方向。
Color:选择颜色。
三、给质粒图谱增加引物序列
1.点击菜单栏Edit→Find(或Ctrl+F),输入引物序列,找到质粒序列对应位置。
2.点击Primers→Addprimer,弹出该界面:

3.按上下游引物选择TopStrand还是BottomStrand,在Primer处可给该引物命名,随后即可显示该引物在图谱Map中的位置,也可以将常用的通用引物建到一个txt文档中,利用importprimer from a list 进行添加。
四、模拟标准限制性克隆
1.打开被插入片段的质粒图谱,选择合适的两个酶切位点,如xbal和EcoRV,点击菜单栏Actions→InsertFragment,点击xbal+Shift键+EcoRV。
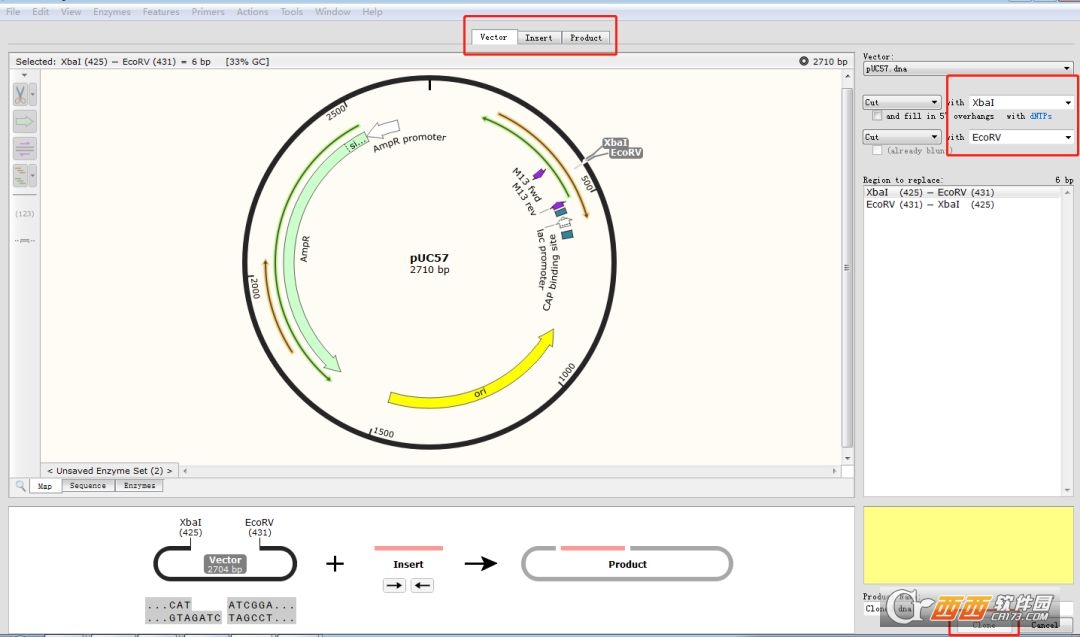
2.点击Insert,在sourceof fragment处选择插入的目的片段来源。

3.点击上述同样的酶,给该重组质粒命名,点击clone。
五、模拟融合克隆
1.打开一个需要插入片段质粒图谱,点击菜单栏Actions→Insert One Fragments,弹出以下窗口:

2.切换到sequence视图,找到想要发生替换的位点。
3.点击Insert,在Source of Fragment处选择拼接或替换片段的另外一个质粒图谱。此时弹出另外一个质粒图谱图样。
4.点击用于替换的片段,观察该片段阅读方向与被替换质粒位点的方向是否一致,如不一致,点击 更换方向。
更换方向。
5.选择后,点击Product,点击Choose Overlapping PCR Primers,此时即形成融合后的质粒图谱。
六、模拟电泳功能
1.点击菜单栏Tools→SimulateAgarose Gel,显示如下界面:

2.点击MW可以选择不同的Marker,其中点击1.2.3.4……分别代表不同的泳道,通过选择不同的酶切位点,模拟各个泳道的电泳结果。
3.以第一泳道为例:点击选中line1,选择BanI、BfaI两个酶切位点,得到模拟电泳结果,如下图:

七、序列比对功能
1.打开一个原始待比对的序列片段,点击菜单栏Tool→Alignwith other Sequences或Alignwith Copied Sequences,如下图所示:

2.在本地文件中将需要比对的序列峰图全部选中,点击打开

3.得到如下比对结果,点击sequence可以看出每个样品的碱基缺失情况:


点击箭头,查看峰图
相比于比如BioXM、Seqman……,SnapeGene在序列比对功能上更加智能,实现了同时比对多种序列和查看峰图等功能。























 大小: 117.1M
大小: 117.1M
 大小: 331M
大小: 331M Overture专业打谱软件5.5.1-7 简体中文版
Overture专业打谱软件5.5.1-7 简体中文版  SPSS 18.0中文版
SPSS 18.0中文版  剪.映windows电脑版2025官方最新版v7.1.0最新专业版
剪.映windows电脑版2025官方最新版v7.1.0最新专业版  MatLab R2012b 特别文件
MatLab R2012b 特别文件  寻星计算程序(卫星天线寻星参数计算)v3.1 绿色版
寻星计算程序(卫星天线寻星参数计算)v3.1 绿色版  八爪鱼采集器v8.58 官方最新版
八爪鱼采集器v8.58 官方最新版  星空抽奖软件无限制版完美注册码版
星空抽奖软件无限制版完美注册码版  利信财务软件专业单机版V6.4免费无限制版
利信财务软件专业单机版V6.4免费无限制版  天学网电脑版v5.6.1.0 官方最新版
天学网电脑版v5.6.1.0 官方最新版  摩客mockplus桌面客户端v3.7.1.0 官方最新版
摩客mockplus桌面客户端v3.7.1.0 官方最新版  淘宝助理天猫版5.3.7.0 官方最新版
淘宝助理天猫版5.3.7.0 官方最新版  易航科技增值税发票电子化软件(增值税发票扫描认证)V2.0免费版
易航科技增值税发票电子化软件(增值税发票扫描认证)V2.0免费版  易房大师v3.2免费版
易房大师v3.2免费版  杰奇小说连载系统免费版
杰奇小说连载系统免费版  发票导出接口软件3.9绿色免费版
发票导出接口软件3.9绿色免费版  发票认证接口软件v2.2绿色版
发票认证接口软件v2.2绿色版  高品智财务公司委托代办项目软件v3.320 官方版
高品智财务公司委托代办项目软件v3.320 官方版  食草族管理专家V2.5.9 绿色免费版
食草族管理专家V2.5.9 绿色免费版